Voici la première proposition de mapping dans le format de ADAM :
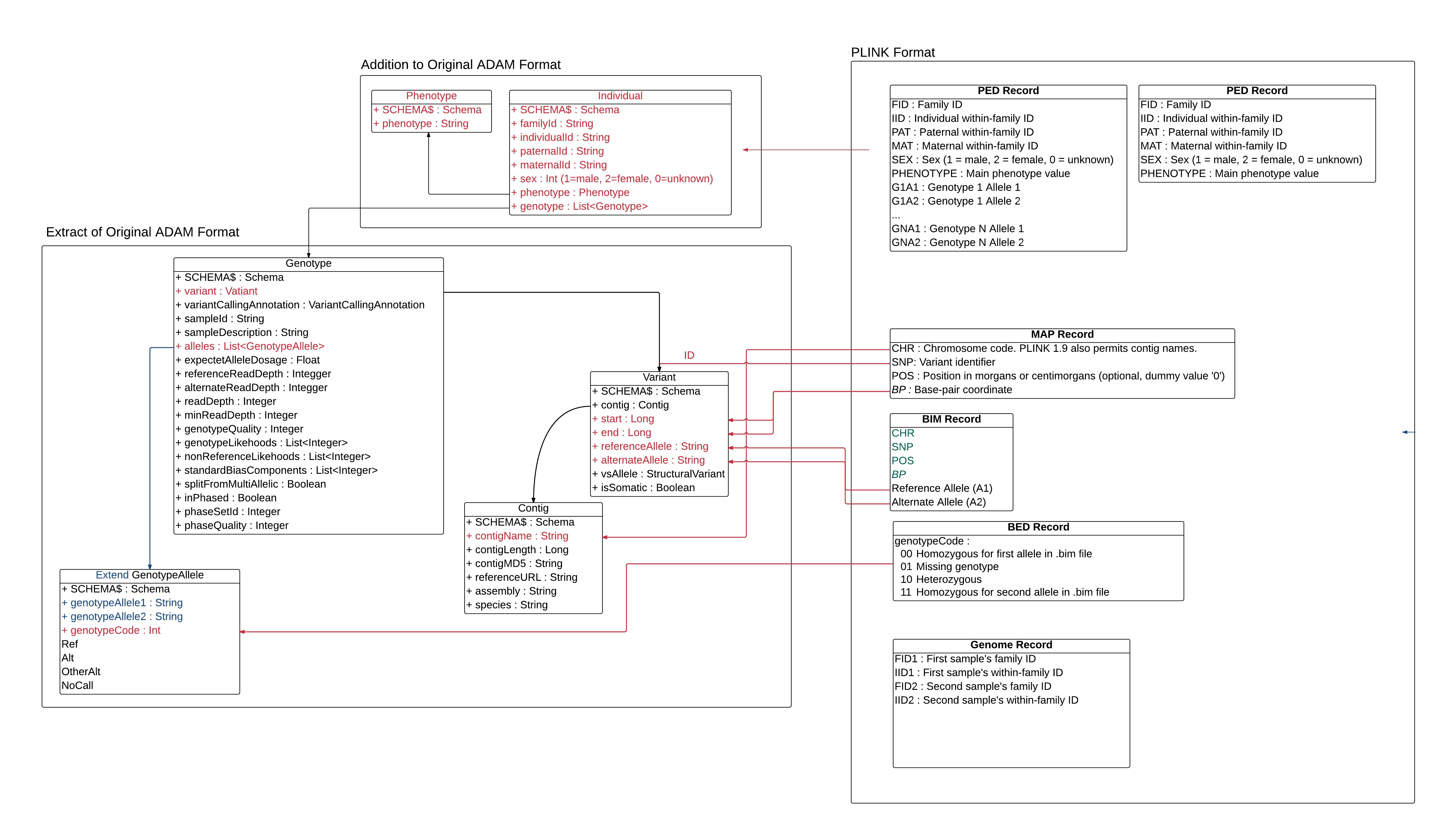
@davidonlaptop, quel est ton avis sur le diagramme ? Pourrai-tu valider/rectifier avant le début d’implémentation ?
Closed iki-v closed 8 years ago
Voici la première proposition de mapping dans le format de ADAM :

@davidonlaptop, quel est ton avis sur le diagramme ? Pourrai-tu valider/rectifier avant le début d’implémentation ?
Suite aux premiers retours, voici la deuxime version :
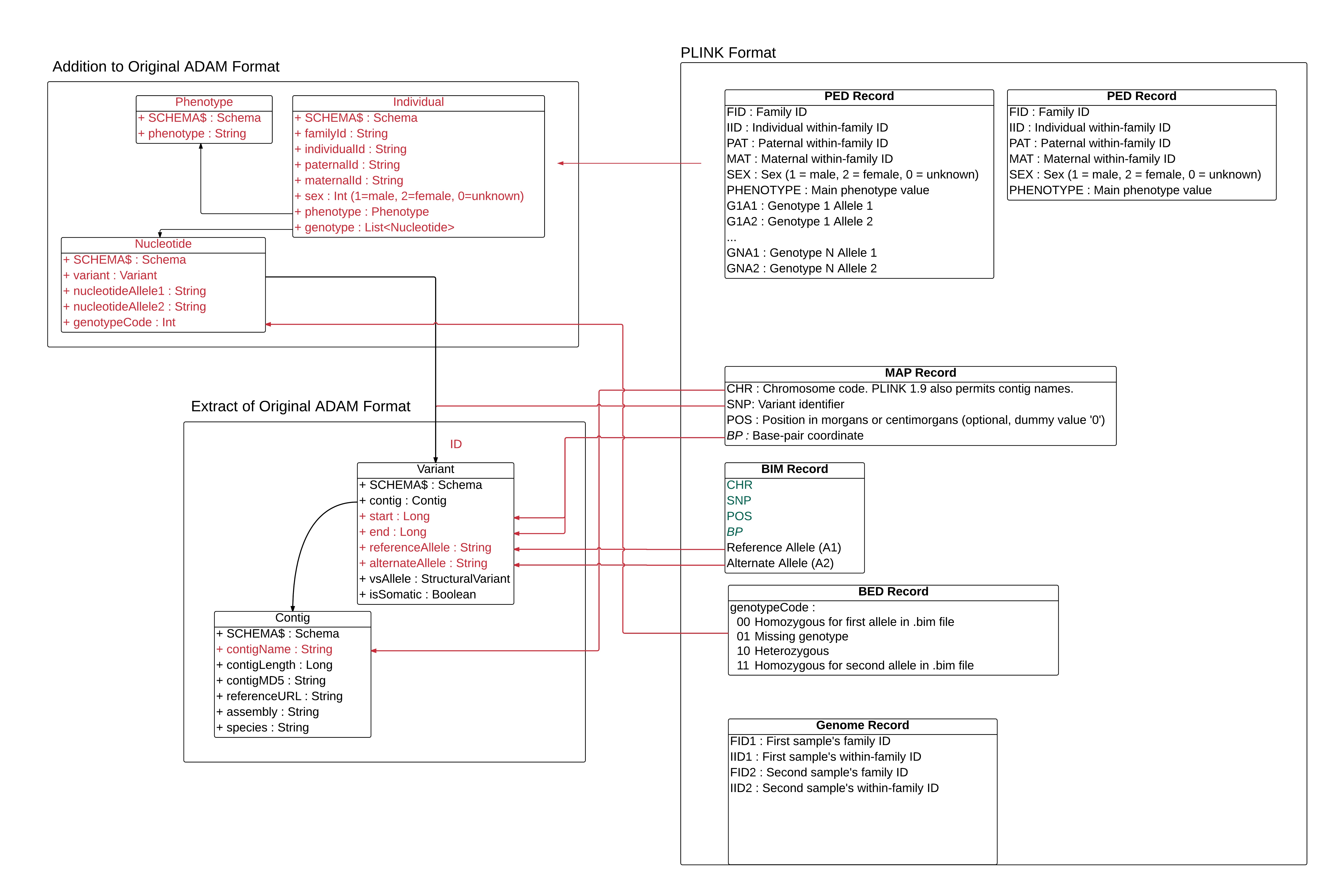
Le GenotypeAllele de Adam est un enum et non une classe, l'équivalent de GenotypeCode ne semble pas present. Peu d'élements se mappent dans le format ADAM original, je propose de simplifier l'ensemble en créant une classe Nucleotide.
@davidonlaptop, pouvons nous commencer l'implementation ?
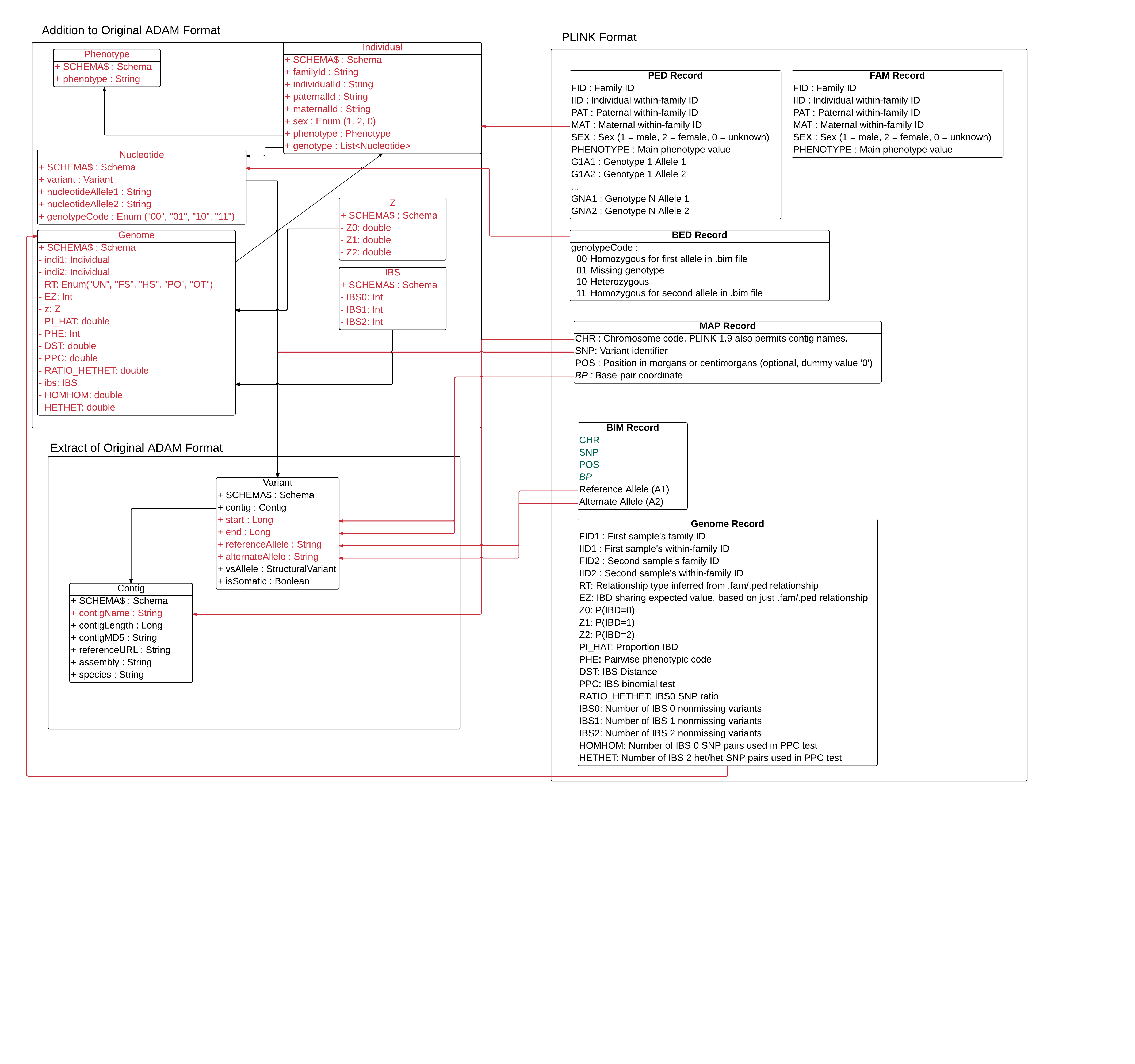
Voici la 3ème version avec le genome:

Il n'y aucune correspondance avec le format ADAM. Ce que je propose c'est d'ajouter une classe "Genome" qui contiendra l'ensemble des attributs définies dans le fichier "Genome record".
@davidonlaptop, nous avons besoin d'un feedback. Merci
For the genome record in ADAM, we should probably nest the Individual record inside: (see image below):
For the gender, please use an Enum and not an integer.
For the Genotype, the relationships does not seem optimal... but I'd have to look at it more in details after my vacation.
As long as it your algorithm produces similar results, you can start coding it.
Voici la version 4 du mapping:
Modification apportée:
Modification apportées:
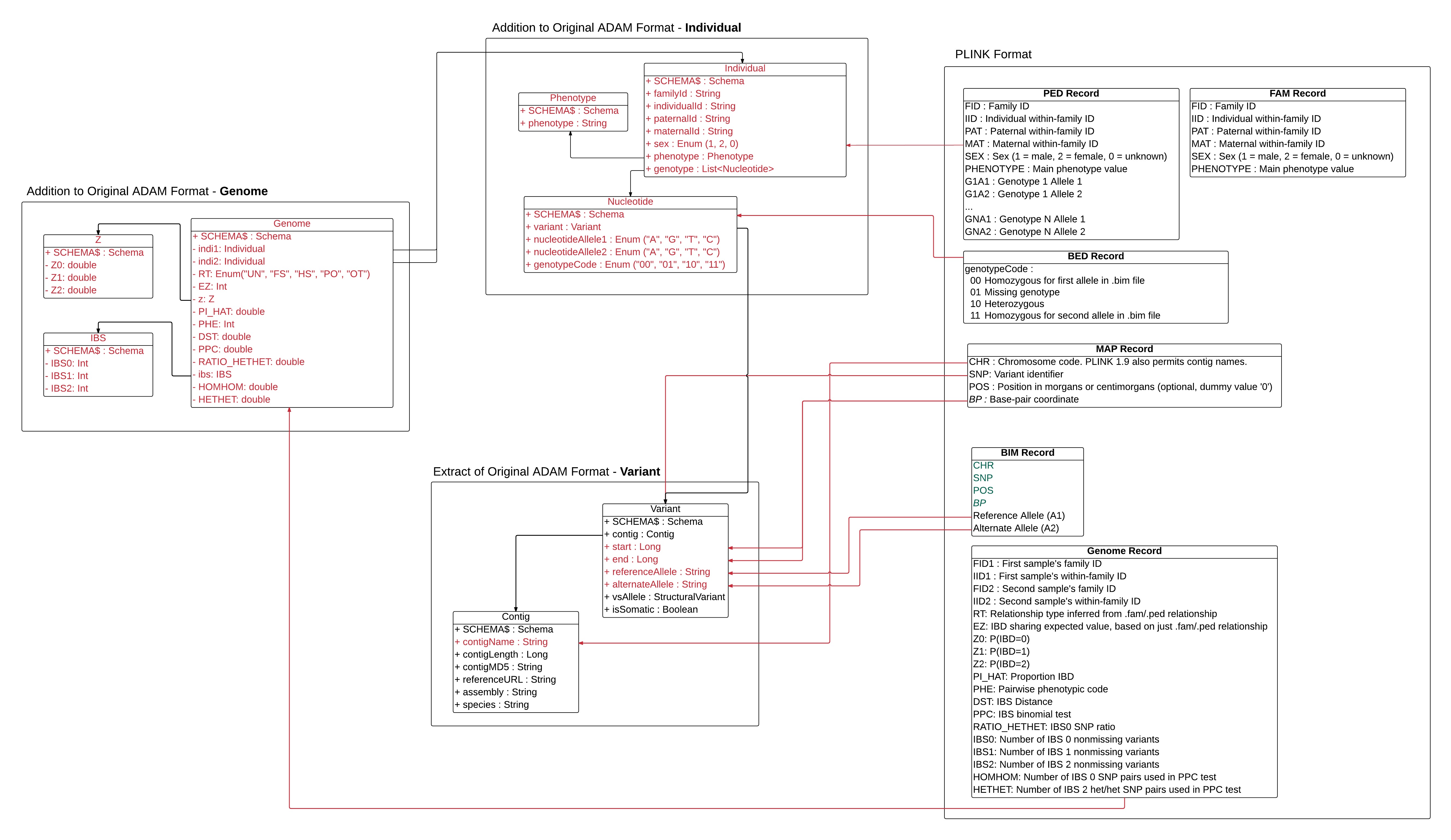
Bonjour @davidonlaptop, as-tu eu le temps de regarder la derniere proposition du modele des donnes ?
Comme convenu hier, les modifications au niveau de Genotype :
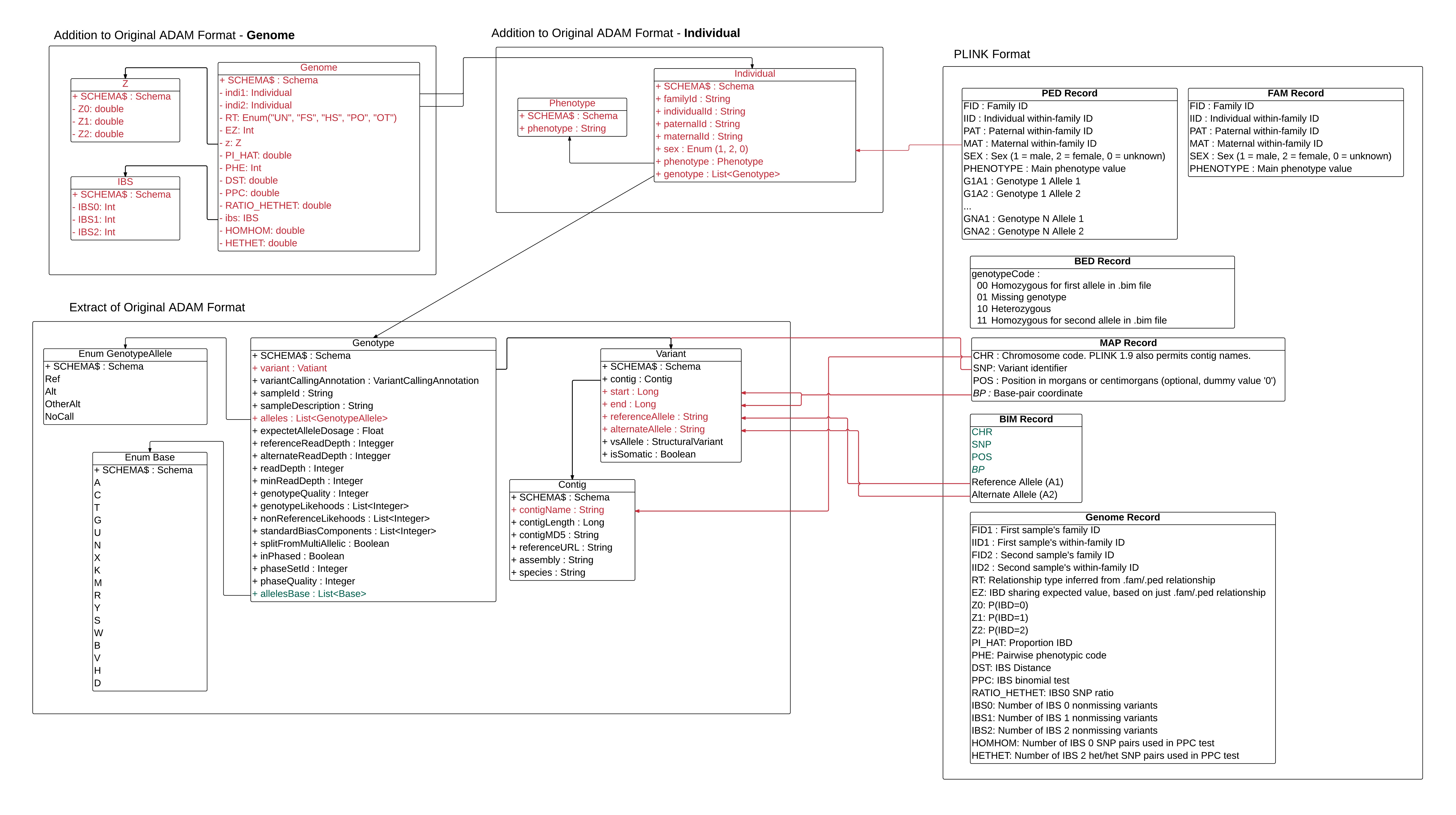
Salut, je croyais que les Z0, Z1, Z2, IBS0, IBS1, IBS2 devaient venir dans le genome, qui aussi devait changer de nom!
Oui tu as raison @Aboubakary, je suis justement en train de faire les modifications par rapport au génome.
Voici les modifications pour le fichier .genome
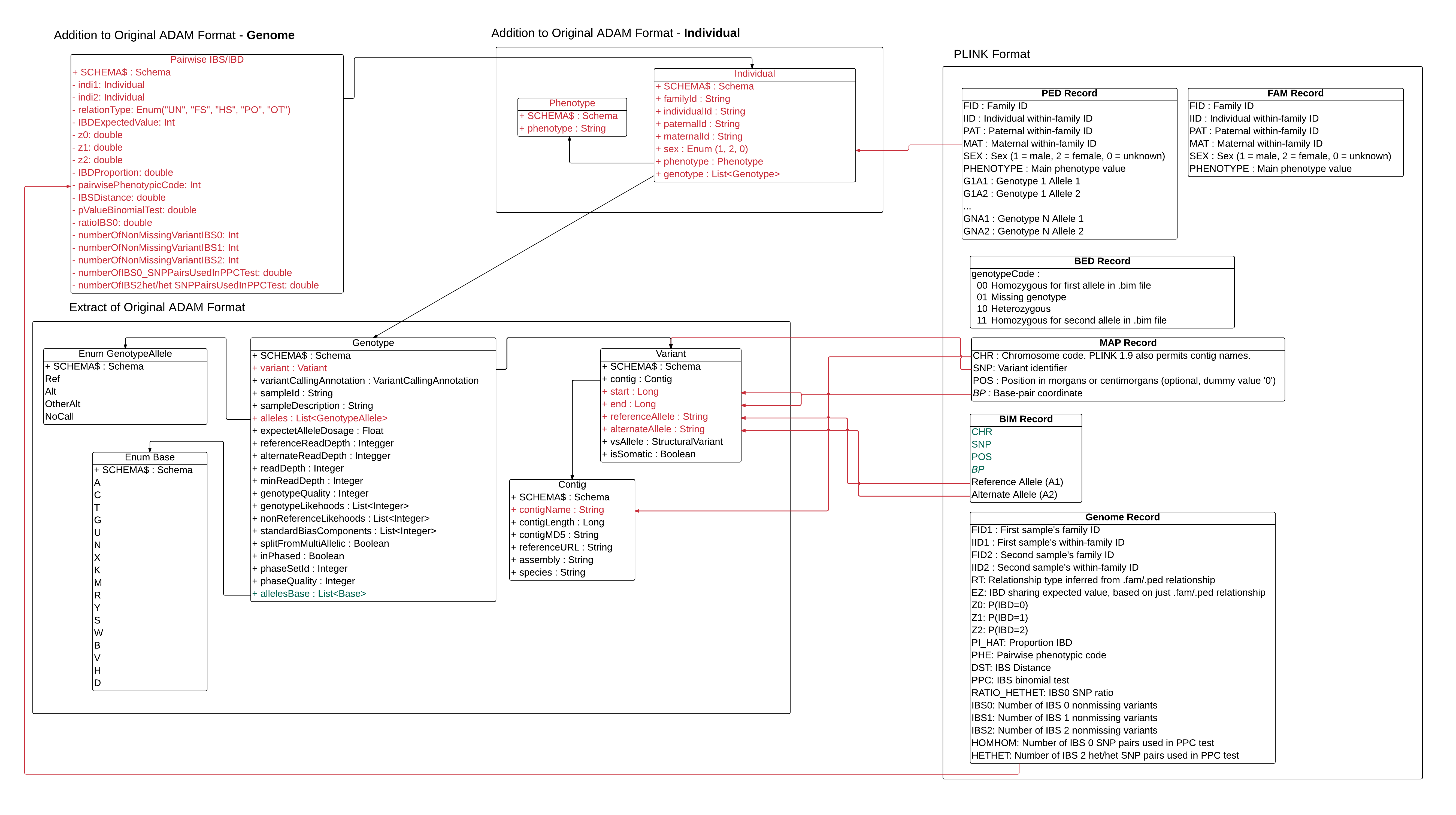
Bonjour @singemanator. Desolé de t'embeter, mais il y a un leger probleme au niveau des noms. Autre le coté metier (ce n'est pas moi qui se potitionne la dessus), c'est la partie technique qui me gene. L'utilisation des " " et "/" n'est pas possible dans le nommage des classses et des attributs. Aussi, il est preferable d'eviter des nos à ralonge (les derniers attributs).
Ce n'est pas un exercice evident de trouver une terminogie parlante et simple dans le domaine de la genetique lorsque nous faisons du logiciel... Comme évoqué hier, nous ne sommes pas les mieux placés pour trouver un nommage metier le plus pertinent possible... Par contre, on se doit de respect les contraintes techniques.
Salut @ikizema, parfait j'en prend, je vais y apporter les modifications nécessaires.
Salut @singemanator , tu souhaite le faire pour quand ? Pour rappel, on s'était éngagé le terminer pour jeudi afin que David puisse le valider...
Voici les modifications pour le fichier .genome sans les ("/") c'était une erreur d'inattention.
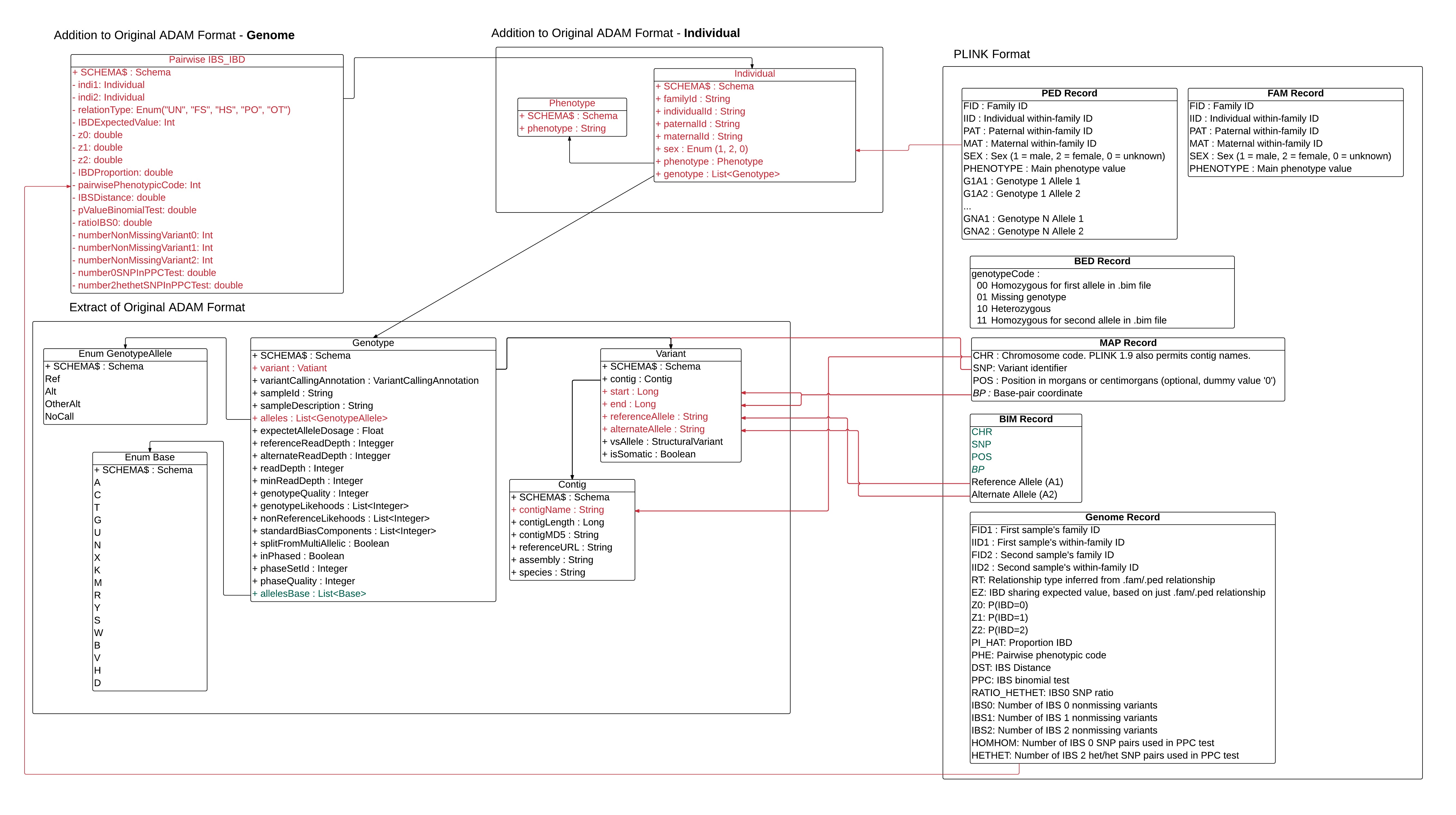
Mais deux questions: 1) Lors de la dernière réunion, j'ai cru comprendre que @davidonlaptop avait dit qu'il ne fallait pas hésiter à mettre de long nom comme ceux qui existe dans un autre schéma. Donc, est-ce que j'ai mal compris? 2) Lorsqu'on fait: Enum("UN", "OT") etc. cela signifie que le Enum est de type String.
J'ai modifié certains noms encore
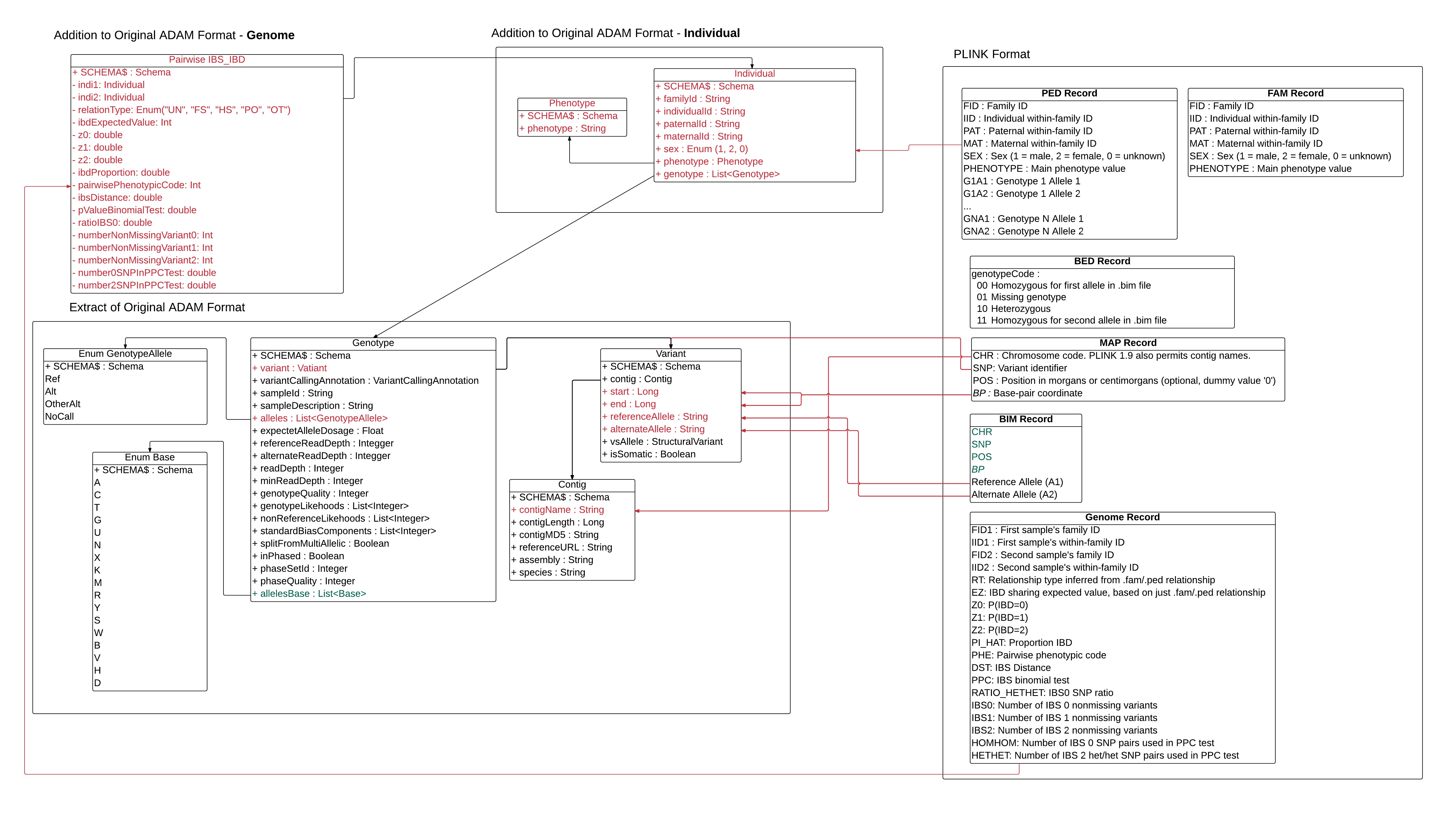
Bonjour @singemanator @ikizema,
Le schéma est beaucoup mieux comme ça!
Quelques suggestions mineures (si vous avez le temps):
Dans Pairwise IBS IBD:
Dans Individual:
Bon travail les gars!
On Sun, Jul 12, 2015 at 7:56 PM, singemanator notifications@github.com wrote:
J'ai modifié certains noms encore [image: image] https://raw.githubusercontent.com/GELOG/adam-ibs/master/WIKI/img/Plink_to_ADAM_Format_Mapping_v9.jpeg
— Reply to this email directly or view it on GitHub https://github.com/GELOG/adam-ibs/issues/43#issuecomment-120774608.
Salut @davidonlaptop, je vais répondre pour ce qui me concerne. En ce qui concerne le phénotype, cela déjà été le cas la semaine dernière et résultait d,un choix réfléchi. Il est vrai que dans les donnes que nous avons, le phenotype possède seulement un string. Cependant, je pense que le besoin peut grandir rapidement, on pourrait imaginer plusieurs paramètres phénotypiques dans la classe... J'ai donc crée une classe a part pour pouvoir compléter les informations au besoin par la suite. Je laisse ainsi ou je l’inclus dans l'attribut d'individu ?
+Beatriz
Je t'accorde qu'un phénotype est bien un concept en soi; mais je ne crois pas qu'il y'a des informations supplémentaires qu'on veuille stocker pour un phénotype. Cela doit rester très flexible car la nomenclature des symptômes (e.g. fièvre, etc.) et ca serait beaucoup de travail de faire un modèle générique pour tous les phénotypes existants. En plus la médecine occidentale est très différente de la médecine indienne ou chinoise.
Selon ce que je comprends, plink permet de mettre un phenotype principal
par individu, ou sinon de spécifier une liste de phénotypes alternatifs
dans un fichier a part. Dans cette optique, je verrais un attribut
"mainPhenotype : String", et un attribut "secondaryPhenotypes :
List
2015-07-14 14:55 GMT-04:00 ikizema notifications@github.com:
Salut @davidonlaptop https://github.com/davidonlaptop, je vais répondre pour ce qui me concerne. En ce qui concerne le phénotype, cela déjà été le cas la semaine dernière et résultait d,un choix réfléchi. Il est vrai que dans les donnes que nous avons, le phenotype possède seulement un string. Cependant, je pense que le besoin peut grandir rapidement, on pourrait imaginer plusieurs paramètres phénotypiques dans la classe... J'ai donc crée une classe a part pour pouvoir compléter les informations au besoin par la suite. Je laisse ainsi ou je l’inclus dans l'attribut d'individu ?
— Reply to this email directly or view it on GitHub https://github.com/GELOG/adam-ibs/issues/43#issuecomment-121340628.
En effet ce serait beaucoup de travail de faire un modèle générique pour tous les phénotypes existants ou a venir. En plus la médecine occidentale est très différente de la médecine indienne ou chinoise.--> Je ne comprends pas le rapport de cette phrase. Qui a parle de medecine chinois ou indienne??? Cependant ceci semble est une solution viable: plusieurs paramètres phénotypiques dans la classe--> qui permettrait de creer les phnotypes et les parametres associes a ce phenotypes, cela donnerait plus de liberte a la personne qui cree ses phenotypes. Donc on aurait la flexibilite pour inclure les phenotypes existants et a venir.Beatriz Sonja Kanzki Bsc BioinformaticsDr Pavel Hamet's LabDr Alain April's team De : David Lauzon davidonlaptop@gmail.com À : GELOG/adam-ibs reply@reply.github.com Cc : GELOG/adam-ibs adam-ibs@noreply.github.com; Beatriz Kanzki beatriz_kanzki@yahoo.ca Envoyé le : mardi 14 juillet 2015 15h56 Objet : Re: [adam-ibs] Mapping des données .ped, .map, .bin, .fam, .bed (#43)
+Beatriz
Je t'accorde qu'un phénotype est bien un concept en soi; mais je ne crois pas qu'il y'a des informations supplémentaires qu'on veuille stocker pour un phénotype. Cela doit rester très flexible car la nomenclature des symptômes (e.g. fièvre, etc.) et ca serait beaucoup de travail de faire un modèle générique pour tous les phénotypes existants. En plus la médecine occidentale est très différente de la médecine indienne ou chinoise.
Selon ce que je comprends, plink permet de mettre un phenotype principal par individu, ou sinon de spécifier une liste de phénotypes alternatifs dans un fichier a part. Dans cette optique, je verrais un attribut "mainPhenotype : String", et un attribut "secondaryPhenotypes : List
2015-07-14 14:55 GMT-04:00 ikizema notifications@github.com:
Salut @davidonlaptop, je vais répondre pour ce qui me concerne. En ce qui concerne le phénotype, cela déjà été le cas la semaine dernière et résultait d,un choix réfléchi. Il est vrai que dans les donnes que nous avons, le phenotype possède seulement un string. Cependant, je pense que le besoin peut grandir rapidement, on pourrait imaginer plusieurs paramètres phénotypiques dans la classe... J'ai donc crée une classe a part pour pouvoir compléter les informations au besoin par la suite. Je laisse ainsi ou je l’inclus dans l'attribut d'individu ?— Reply to this email directly or view it on GitHub.
L'objectif de cette tache est de proposer un mapping de format de données Plink dans le Format ADAM.
Proposition de mapping pour le extensins .ped, .map, .bim, .fam, .bed.