非常感谢明哥一次次的精心准备与分享!!!
Open Ming-Lian opened 5 years ago
非常感谢明哥一次次的精心准备与分享!!!

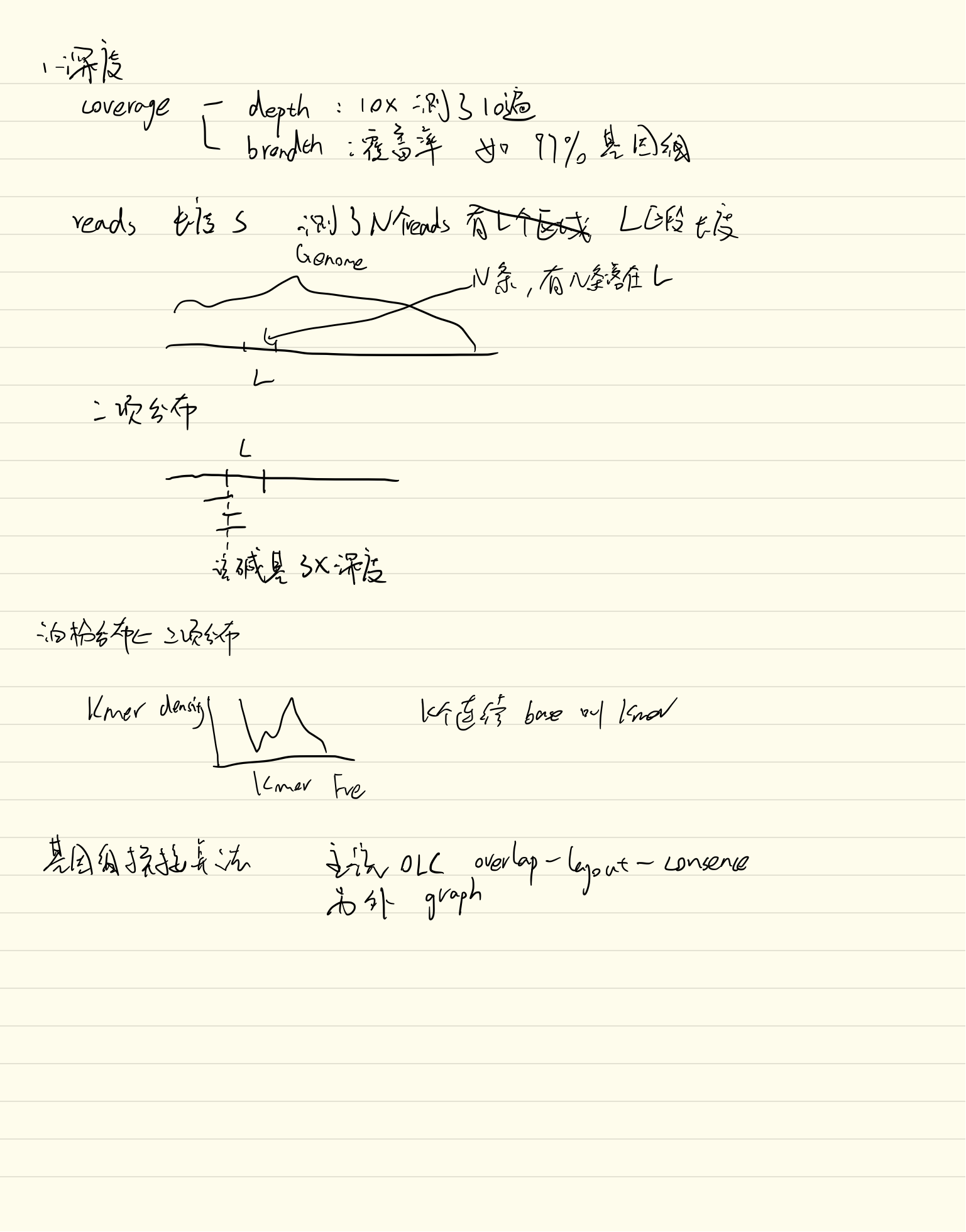
1.1.1用泊松分布建模测序深度与测序覆盖度的关系 设定序列长度为G,读数数目为N,读长为S,测序片段数目为N,落在L中的序列数目为D,D符合重复N次的,平均值为L/G的二项分布,D~(N,L/G)。 若设定L=S,则D=最后一位碱基覆盖的次数(假定序列的起始点落在该区域视为序列落在该区域),则平均值为S/G。当二项分布N→无穷,概率S/G→0。
测序深度 每个碱基被测到的次数 测序覆盖率 被测到至少1次的碱基的数目和全部碱基数目的比值,可以视为任一碱基位点覆盖的概率。
1.1.2用Kmer图评估基因组的重复率和杂合率 按照Kmer频率为横坐标,密度为纵坐标作图。 K-mer的频率=L-K+1,当中间的峰两侧(频率为2倍的位置)有峰,则证明有重复或者杂合。
1.1.3基因组拼接算法:de Bruijn图的构建与搜索欧拉路径 拼接算法:1OLC overlap;2graph。
图论的问题是有节点有边的问题,如七桥问题,如何找到欧氏路径:每座桥只走一次,最后回到出发点。 算法:随机游走的蚂蚁。1随机取得起始点(如果选的不好容易形成局部最优),确认一条欧氏路径2从原来的欧氏路径中包含其它分支路径的点之中,随机选择一个,再次寻找欧氏路径。3不断重复前两步,直到找到最好的欧氏路径。 1.1.4 根据mapping结果,确定突变位点的碱基。 设定D为比对结果,g为实际基因集。设定g1为一致纯合,设定g2为不一致纯合,g3为不一致杂合。 max[P(g1|D);P(g2|D)], P(g1|D)=连乘K次(对的概率)连乘L次(错的概率) L:reads总数;k:与参考序列一致的reads数;L-k:与参考序列不一致的reads数;m:倍性(人类为2倍体)。 A的概率=连乘(P(测得对)+P(测得错))=(1-ε)g/m+εg/m。
对于人的单个reads来说,Assuming that sequencing error rate is 0.01, 假如参考是C,如果我们现在看到一条reads是C在某个位点 那么 P(reads|A/A, read mapped) = 0.01 P(reads|A/C, read mapped) = 0.50 P(reads|C/C, read mapped) = 0.09
https://ming-lian.github.io/2019/09/03/Bioinfo-ML-Club-7th/